摘 要:目的 探讨DNA甲基化修饰在口腔白斑癌变中的作用。方法 选取40例口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)组织和28例口腔白斑(oral leukoplakia,OLK)组织以及40例健康口腔黏膜组织进行DNA甲基化检测。下载GEO(Gene Expression Omnibus)数据库中OSCC、OLK和健康口腔黏膜的表达谱数据。分别对DNA甲基化数据和表达谱数据进行重复性比对,对DNA甲基化数据进行差异分析以及对相应表达谱数据进行IPA分析(Ingenuity Pathway Analysis)。结果 数据分析结果显示DNA甲基化具有更大的灵活性和不稳定性。IPA通路分析表明,OLK差异甲基化位点相关基因主要富集在细胞运动和分化过程。OSCC差异甲基化位点相关基因主要富集在细胞增殖、迁移、氧化调节、抗凋亡等过程。OLK和OSCC差异甲基化位点相关基因共同富集于磷酸肌醇代谢过程和磷脂酶C信号通路。结论 DNA甲基化改变参与口腔鳞癌形成,磷酸肌醇代谢激活可能促使口腔白斑癌变。
关键词:口腔白斑;口腔鳞状细胞癌;表观遗传学; DNA甲基化;肌醇;磷酸肌醇;代谢;磷脂酶C;炎症;钙信号;
Significance of phosphoinositol metabolism by DNA methylation may contribute in oral
leukoplakia carcinogenesis
WANG Guangchao
LIU Ljun JIANG Weiwen
Department of Oral Mucosal Diseases, Shanghai Ninth People's Hospital, College of Stomatology,
Shanghai Jiao Tong University School of Medicine, National Clinical Research Center for Oral
Diseases, Shanghai Key Laboratory of Stomatology & Shanghai Research Institute of Stomatology
Abstract:Objective To study the role of DNA methylation in oral leukoplakia carcinogenesis.Methods DNA methylation was detected in forty cases of oral squamous cell carcinoma(OSCC),twenty-eight cases of oral leukoplakia(OLK) and forty cases of healthy oral mucosa.Download the expression profile data of OSCC,OLK and healthy oral mucosa from Gene Expression Omnibus(GEO) database.DNA methylation data and expression profile data were compared for repeatability,DNA methylation data for difference analysis and corresponding expression profile data for IPA pathway analysis.Results The data analysis showed that DNA methylation had greater flexibility and instability.Ingenuity Pathway Analysis(IPA) analysis showed that genes related to OLK differential methylation sites were mainly concentrated in the process of cell movement and differentiation.Genes related to differential methylation sites of OSCC are mainly enriched in cell proliferation,migration,oxidation regulation,and anti-apoptosis processes.The genes associated with OLK and OSCC differential methylation sites are co-enriched in phosphoinositol metabolism and phospholipase C signaling pathway.Conclusion DNA methylation is involved in the formation of oral squamous cell carcinoma,and the activation of phosphoinositol metabolism may promote oral leukinoma.
Keyword:oral leukoplakia; oral squamous cell carcinoma; epigenetics; DNA methylation; inositol; phosphoinositol; metabolism; phospholipase C; inflammation; calcium signal;
口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)是常见的恶性肿瘤之一,吸烟和酗酒是OS-CC的主要病因[1]。口腔白斑(oral leukoplakia,OLK)是口腔黏膜上以白色斑块或斑片为主的损害,不能定义为其它任何疾病,是最常见的口腔癌前病损。OLK恶变的最可预测因素是活检中存在异型增生,研究表明异型增生患者的恶性风险为31.4%~36.3%[2]。因此,明确OLK和OSCC的生物学关系,对于预防OLK癌变和OSCC的早期诊断有重要意义。
除基因组突变外,表观遗传学改变已被确定为OSCC的重要特征[3],尤其是转录起始位点启动子附近发生的DNA甲基化,通常会造成基因表达沉默和信号通路失调[4]。而且,在许多癌症中,通过启动子DNA甲基化导致的抑癌基因失活甚至比体细胞突变更加频繁[5]。DNA甲基化可能推动肿瘤发生和恶性进展。本文探讨DNA甲基化修饰在口腔白斑癌变中的作用,为揭示DNA甲基化在癌前病变和肿瘤发病过程中的关键调控因子提供依据。
1 材料和方法
1.1 收集样本和Illumina 450K甲基化芯片检测
在上海交通大学医学院附属第九人民医院口腔颌面外科和口腔黏膜病科就诊患者中收集40例OSCC组织样本、28例OLK组织样本和40例正常健康口腔黏膜组织样本。其中,40位捐赠OSCC组织样本的患者来自口腔颌面外科,口腔黏膜病史不详。本研究已获得上海交通大学医学院附属第九人民医院伦理审查委员会的批准,并获得了患者的知情同意。使用Illumina 450K甲基化芯片(博奥生物,北京)检测上述样本,得到DNA甲基化位点的β值数据[6]。
1.2 GEO数据库中表达谱数据整理
检索GEO(Gene Expression Omnibus,GEO)数据库,得到OSCC、OLK以及正常口腔黏膜组织的表达谱数据。根据实验需求选用GSE33205[7]和GSE85195[8]两个表达谱数据集。
1.3 DNA甲基化谱和表达谱数据特点统计分析
在R语言平台上,分别在40例OSCC组织样本、28例OLK组织样本以及40例健康口腔黏膜组织样本中随机选取1例OSCC、1例OLK和1例健康口腔黏膜样本,选取DNA甲基化β值最少的5%位点。计算这些位点在上述3例不同样本之间的重叠率。重复上述过程10次,得到不同类别样本的平均重叠率。分别在40例OSCC组织样本、28例OLK组织样本以及40例健康口腔黏膜组织样本中随机选择2例相同类别的样本,重复上述过程10次,得到相同类别样本的平均重叠率。下载GSE85195数据集,分别随机选取1例OSCC样本,1例OLK样本以及另外1例健康口腔黏膜样本,得到表达值位于95%以上的探针ID。计算这些探针ID在不同样本之间的重叠率。重复上述过程10次,得到不同类别样本的平均重叠率。在GSE85195数据集中随机选取2例OSCC,2例OLK,在GSE33205数据集中随机选择2例健康口腔黏膜样本,得到表达值位于95%以上的探针ID,计算这些探针在相同类别样本中的重叠率。重复上述过程10次,得到相同类别样本的平均重叠率。
1.4 筛选OSCC和OLK差异DNA甲基化位点并整理相应的表达谱数据
在R语言平台上,所有样本的β值转换为M值[9]。分别计算OSCC、OLK和健康口腔黏膜每个位点的平均M值。选取OSCC vs.Normal平均值相差大于1.4的位点,以及OLK vs.Normal平均值相差大于1.4的位点。加载Limma包,对上述位点进行差异分析[10]。选取P<0.01且校正后P<0.05的位点。整理后得到OSCC差异甲基化位点和OLK差异甲基化位点。下载GSE85195的数据集,提取OSCC和OLK差异甲基化位点所在基因的表达谱数据。计算OSCC和OLK每一个探针的平均表达强度,输出整理后的表格供后续分析使用。
1.5 生物学通路分析
在IPA生物学通路(Ingenuity Pathway Analysis)平台(QIAGEN,德国)上,提交OSCC和OLK差异甲基化位点相关基因的表达谱数据。分别进行核心分析和对比分析。得到分析报告后,分别下载经典通路的富集结果以及显著富集的网络。在对比分析中下载共同富集的通路。
2 结果
2.1 DNA甲基化谱相对表达谱数据具有更多的差异性
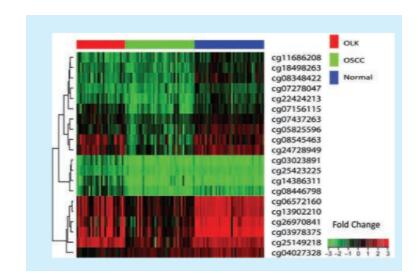
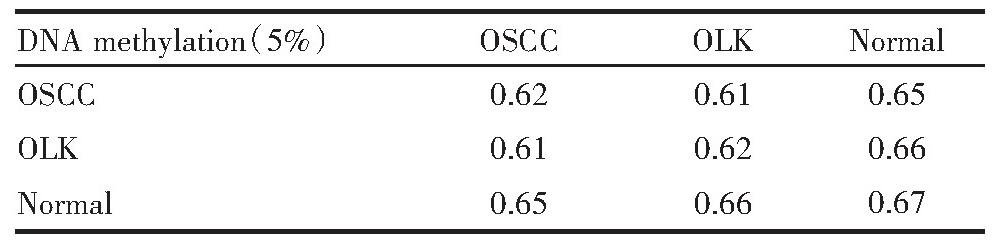
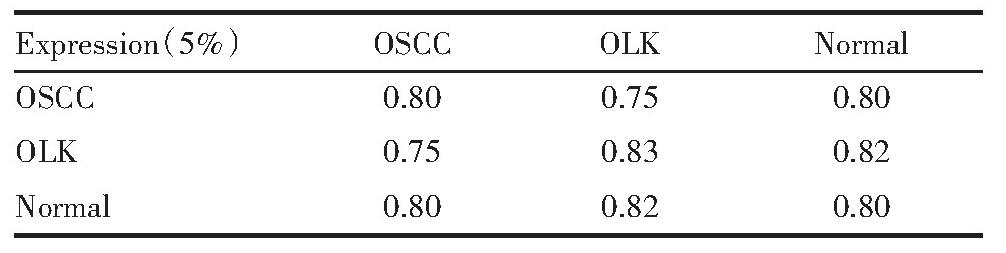
本实验共获得48 5203个DNA甲基化位点在28例OLK,40例OSCC和40例健康口腔黏膜样本中的β值数据(图1)。GSE33205数据集中含有50例HNSCC和25例健康口腔黏膜样本。GSE85195数据集中含有15例OLK和34例OSCC和1例健康口腔黏膜样本(表1)。在各样本两两比较中,DNA甲基化谱的数据在贝塔值最小的5%重叠率为61%~67%,而表达谱数据在表达量最多的5%重叠率为75%~83%(表2、表3)。
2.2 差异DNA甲基化位点和表达谱数据整理结果
本实验在28例OLK和40例健康口腔黏膜DNA甲基化芯片数据比对中得到4 396个OLK差异DNA甲基化位点。在40例OSCC和40例健康口腔黏膜DNA甲基化芯片数据比对中得到3 475个OSCC差异DNA甲基化位点。其中,OLK差异DNA甲基化位点中有671个DNA甲基化位点在OLK组织中上调,有3 725个DNA甲基化位点在OLK组织中下调。另外,在OSCC差异DNA甲基化位点中有2 768个DNA甲基化位点在OSCC组织中上调,有707个DNA甲基化位点在OSCC组织中下调。OLK和OSCC差异DNA甲基化位点相交423个。4 396个OLK差异DNA甲基化位点经注释得到1 809个对应基因。3 475个OSCC差异DAN甲基化位点经注释得到1 457个对应基因。其中,这两者相交338个基因。提取GSE85195数据集的表达谱数据,1 809个OLK差异DNA甲基化位点相关基因位于表达谱芯片中的2 862个探针上,其中1 805个探针在OLK表达谱数据中显示上调,1 057个探针在OLK表达谱数据中显示下调。1 457个OSCC差异DNA甲基化位点相关基因位于表达谱芯片中的2 198个探针上,其中1 117个探针在OSCC表达谱数据中显示上调,1 081个探针在OSCC表达谱数据中显示下调。
图1 Illumina 450K甲基化芯片部分检测热图
表1 本次实验选用的外部表达谱数据集
表2 DNA甲基化谱在β值最小时的5%重叠率
表3 表达谱在表达量最大时的5%重叠率
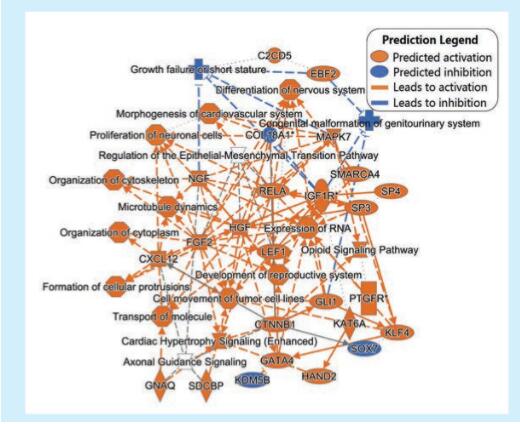
2.3 OLK差异DNA甲基化相关基因主要参与细胞运动和分化
OLK差异DNA甲基化相关基因的IPA生物学通路和网络分析表明,在OLK组织中,细胞骨架的组织、多种发育过程、结缔组织分化和细胞运动等过程处于活跃状态。其中Sp3转录因子(Sp3 transcription factor,SP3)、Sp4转录因子(Sp4 transcription factor,SP4)、淋巴增强因子1(lymphoid enhancer binding factor 1,LEF1)、KLF4(Kruppel like factor4)、早期B淋巴细胞因子(early B-cell factor 2,EBF2)、GATA结合蛋白4(GATA binding protein 4,GATA4)、GLI1(GLI family zinc finger 1,GLI1)活化,激活RNA转录和表达。细胞粘附分子SDCBP(syndecan binding protein,SDCBP)、CTNNB1(catenin beta 1,CTNNB1)活化促进细胞运动和分化。肝细胞生长因子(hepatocyte growth factor,HGF)、神经生长因子(nerve growth factor,NGF)、成纤维细胞生长因子2(fibroblast growth factor 2,FGF2)和胰岛素样生长因子1受体(insulin-like growth factor 1 receptor,IGF1R)以及丝裂原活化激酶-7(mitogen-activated protein kinase,MAPK7)活化增强细胞存活抗凋亡。同时,染色质修饰蛋白赖氨酸特异性脱甲基酶5B(lysine demethylase 5B,KDM5B)抑制,赖氨酸乙酰转移酶6A (lysine acetyltransferase 6A,KAT6A)、SWI/SNF相关,基质关联,肌动蛋白依赖染色质调控因子,亚家族a,成员4(SWI/SNF related,matrix associated,actin dependent regulator of chromatin,subfamily a,member 4,SMARCA4)活化使得组蛋白和染色质结构得到相应修饰。含钙离子依赖性C2结构域的蛋白质5(C2 calcium dependent domain containing 5,C2CD5)活化促进葡萄糖转运(图2)。
2.4 OSCC差异DNA甲基化相关基因参与细胞癌变的多个过程
在IPA生物学通路分析平台上,得到OSCC差异甲基化相关基因相互作用网络富集结果,富集评分大于或等于40的网络共有3个。网络1相关基因主要富集在细胞运动,迁移,分化等生物学过程和通路。网络2相关基因主要富集在发育,氧化调节、蛋白折叠、抗凋亡等生物学过程和通路。网络3相关基因主要富集在离子转运,细胞迁移,细胞周期等生物学过程和通路。为了方便展示,把这三个网络合并为一个网络(图3)。其中,视黄醇脱氢酶(dehydrogenase/reductase 3,DHRS3)、谷胱甘肽过氧化物酶3(glutathione peroxidase 3,GPX3)、细胞色素P450家族成员CYP26A1(cytochrome P450 family 26 subfamily A member 1,CYP26A1)以及多种氧化损伤保护蛋白下调,从而增加细胞氧化损伤,进一步加重蛋白折叠错误率。在血管内皮细胞生长因子(vascular endothelial growth factor,VEGF)的作用下,多种同型盒基因表达改变促进细胞增殖和分化。丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)信号通路活化促使肌钙蛋白和锚定蛋白激活,进而重组细胞骨架。髓系细胞触发受体2(triggering receptor expressed on myeloid cells 2,TREM2)活化触发组成型炎性细胞因子的产生形成慢性炎症,伽马分泌酶(Secretaseγ)增多使线粒体功能失调,钾通道四聚化结构域-2(potassium channel tetramerization domain containing 2,KCTD2)、氯离子通道Tweety家族蛋白成员TTYH1(Tweety family member 1)、钙离子通道兰尼定受体1(ryanodine receptor 1,RYR1)以及钠离子和碳酸根离子转运蛋白SLC4A7(solute carrier family 4 member 7)的表达也受到影响,这些改变共同促进细胞增殖,迁移和染色质重塑。
2.5 OSCC和OLK差异DNA甲基化位点相关基因共同富集在磷酸肌醇代谢过程
在IPA生物学通路分析平台上,对比分析结果表明,OSCC和OLK差异甲基化位点相关基因在多种磷酸肌醇生物合成和代谢以及磷脂酶C信号通路富集(表4)。其中肌醇磷酸生物合成,3-磷酸肌醇生物合成,肌醇四磷酸磷酸酯生物合成以及5-磷酸肌醇代谢在OLK和OSCC中均处于活化状态。磷脂酶C信号通路在OSCC中抑制,在OLK中活化。
图2 口腔白斑差异DNA甲基化位点相关基因主要参与细胞运动和分化
图3 口腔鳞状细胞癌差异DNA甲基化相关基因富集网络
3 讨论
为了验证在肿瘤形成过程中DNA甲基化是否具有不同程度的改变,本实验选用DNA甲基化程度最低的位点进行重复比对分析。低表达的m RNA在实验过程中不稳定,并且在检测过程中容易受系统误差干扰,因此表达谱对比部分,本实验选用表达量最高的5%进行分析。研究表明,DNA甲基化修饰具有灵活变化的特点,表达谱相对稳定。由于基因表达受到多种表观因素的影响,表观遗传的可塑性使得细胞可以通过启动新的转录程序增加调节潜力,从而改变细胞的可延展方式[11]。这意味着表观系统具有更早的接受外部信息的能力,可以被应用于肿瘤形成的早期诊断。
肿瘤发生和进展是涉及多层次、多过程、多通路的复杂过程。研究表明,肿瘤的发生伴随着多个基因突变以及染色体结构异常,从而促使细胞进入恶性增殖过程[12]。DNA甲基化是重要的染色质修饰手段。DNA通过甲基化修饰,可以沉默或激活相关基因,改变染色体折叠状态,进而促进机体发育和细胞分化[13]。目前已有大量研究表明,DNA甲基化修饰的改变在OSCC发病过程中有重要作用,尤其是抑癌基因启动子区域高甲基化失活可能成为细胞癌变的关键步骤[3,14]。本研究差异DNA甲基化结果证实,不仅仅在肿瘤发生阶段,甚至在癌前病变中,就已经存在较多DNA甲基化的改变。研究表明,在癌前状态下,由于外部不良刺激以及局部炎症压力,促使细胞骨架重组和上皮间充质转变[15]。在这个过程中染色质也会有相应的重塑,而DNA甲基化修饰是重要的调节方式[16]。
在肿瘤发生早期,由于内外压力共同作用,细胞会有一定时间的呼吸爆发,产生过量的反应性活性氧。这些活性氧产物会通过各种途径导致线粒体呼吸链损伤以及DNA突变。维持DNA的高保真性是一个高耗能的过程。为了维持DNA稳定,在线粒体功能不足的情况下,那些选择无氧糖酵解的细胞有更大的可能性从凋亡的命运中逃离出来。在OSCC生物学通路分析中,伴随DNA甲基化改变,多个抗氧化保护蛋白下调。它们有的定位于线粒体,有的定位于细胞质。本课题组以往的研究发现,HNSCC中的泛醌还原酶亚基A13处于高甲基化低表达状态[17]。与此类似,本研究OSCC表达谱结果证实泛醌还原酶亚基A1在OSCC中同样处于低表达状态。有趣的是,泛醌还原酶亚基A1在OLK中处于低甲基化高表达状态。泛醌还原酶位于线粒体内膜,是有氧氧化呼吸链的重要蛋白。以上研究结果反映OSCC发生过程中可能存在有氧呼吸增强而后弱化的过程,这为抗氧化药物预防OLK癌变提供了依据。
多个离子通道和转运分子活化结果表明OSCC进展过程中伴随频繁的离子内外交换,可能参与多个癌变过程。同以往研究结果一致,在OSCC和OLK组织中,钙通道蛋白兰尼定受体2(ryanodine receptor 2,RYR2)处于高甲基化低表达状态[18]。另外,本实验发现OSCC和OLK组织中的RYR1处于低甲基化高表达状态。钙离子信号在分化和组织动态平衡中具有重要作用。由于体细胞突变或表观遗传沉默引起的钙离子信号变化改变会削弱分化,与其它致癌因素相结合,可能加速促进口腔黏膜细胞癌变的进程。
炎细胞浸润和免疫反应是细胞癌变的常见现象。持续的炎性环境产生了大量的活性氧簇,促使炎细胞释放细胞因子、趋化因子、生长因子等炎性介质,其引发的级联反应能够诱导细胞增殖。中性粒细胞和巨噬细胞活化后将产生大量反应性活性氧和反应性活性氮等氧化物。它们可促进增殖细胞发生DNA损伤、癌基因激活及抑癌基因失活。反复的慢性炎症刺激还将通过各种信号通路募集免疫抑制性细胞,这些细胞激活后能抑制T细胞和自然杀伤细胞的功能,促进突变细胞免疫逃逸。在炎症反应中,磷酸肌醇代谢参与多种炎症信号转导[19]。本研究证实,从癌前状态的OLK到肿瘤形成的OSCC均伴随磷酸肌醇代谢活动的增强,这提示了DNA甲基化状态改变导致磷酸肌醇代谢活动增强可能是OSCC发生发展的重要诱因;磷酸肌醇代谢以及磷脂酶C相关基因表达和DNA甲基化检测可能成为OSCC风险评估的重要因素。
【Author contributions】Wang GC collected,processed and analyzed the data and wrote the article.Liu LJ processed the research.Jiang WW designed the study and reviewed the article.All authors read and approved the final manuscript as submitted.
参考文献
[1] Bewley AF,Farwell DG. Oral leukoplakia and oral cavity squamous cell carcinoma[J] Clin Dermatol,2017,35(5):461-467.doi:10.1016/j clindermat01.2017.06 008.
[2] Mehanna HM.Rattay T,Smith J,et al.Treatment and follow-up of oral dysplasia-a systematic review and meta-analysis[J].Head Neck,2009,31(12)-1600-1609 doi:10. 1002/hed 21131.
[3] Hema KN,Smitha T,Sheethal HS ,et al. Epigenetics in oral squamous cell carcinoma[J]J Oral Maxillofac Pathol,2017,21(2):252-259.doi:10.4103/jomfp.JOMFP. _150_ 17.
[4] Zhang J.Yang C.Wu C.et al DNA methyltransferases in cancer.biology.paradox aberrations ,and targeted therapy[J]. Cancers (Basel),2020.12(8):2123. dol:10 3390/cancers12082123.
[5] Liu X.Zhang T,Li Y,et al.The role of methylation in the Cp G island of the ARHI promoter region in cancers[J].Adv Exp Med Biol,2020,1255.123-132. doi:10. 1007/978-981-15-4494-1_ 10.
[6] Chatterjee A,Rodger EJ,Morison IM.et al. Tools and strategies for analysis of Genome Wide and Gene- Specific DNA methylation patterns[J].Methods Mol Biol,2017,1537:249-277 doi:10.1007/978-1-4939-6685-1. _15.
[7] Stansfield JC ,Rusay M,Shan R.et al. Toward Signaling-Driven biomarkers immune to normal tissue contamination[J].Cancer Inform,2016,15:15-21.doi:10.4137/CIN. S32468.
[8] Bhosale PG,Cristea S,Ambatipudi S,et al.Chromosomal alterations and gene expression changes associated with the progression of leukoplakia to advanced gingivobuccal cancer[J]. Transl Oncol,2017,10(3):396 409 doi:10. 1016/j. tranon.2017.03 008.
[9] Du P,Zhang X.Huang CC.et al.Comparison of beta-value and mvalue methods for quantifying methylation levels by microarray analysis[J].BMC Bioinformatics,2010.11(1):587 doi:10.1186/1471-2105-11-587.
[10] Ritchie ME,Phipson B.Wu D.et al.Limma powers difterential expression analyses for RNA-sequencing and microarray studies[J] Nucleic Acids Res,2015,43(7):e47 .doi: 10.1093/nar/gkv007.
[11] Eckersley-Maslin M.Keeping your options open:insights from Dppa2/4 into how epigenetic priming factors promote cell plasticity[J]. BiochemSoc Trans ,2020,48(6):2891 -2902 doi:10. 1042/BST20200873.
[12] Oleksiewicz U,Machnik M. Causes ,ffects, and clinical implications of perturbed patterns within the cancer epigenome[J] Semin Cancer Biol,2020:S1044-579X(20)30274. doi:10.1016/j .semcancer. 2020.12.014.
[13] Wang J.Yang J,Li D.et al. Technologies for targeting DNA methylation modifications basic mechanism and potential application in cancer[J].Biochim Biophys Acta Rev Cancer,2021,1875(1);188454. doi:10.1016/. bbcan 2020 188454.
[14] Romanowska K Sobecka A. Rawuszko-Wieczorek AA.et al.Head and neck squamous cell carcinoma:epigenetic landscape[J]. Diagnostics (Base),2020.11(1):34 doi: 10 3390/diagnostics11010034.
[15] Arnold L,Enders J,Thomas SM.Activated HGF-C-Met axis in head and neck cancer[J] Cancers (Basel),2017.9(12):169.doi:10. 3390/cancers9120169.
[16] Abe M.Yamashita S,Mori Y,et al.High-isk oral leukoplakia is associated with aberrant promoter methylation of multiple genes[J] BMC Cancer,2016,16(1):350. doi:10.1186/s12885-016-2371-5.
[17] Zhang XY,Li M,Sun K.et al. Decreased expression of GRIM-19by DNA hypermethylation promotes aerobic glycolysis and cell proliferation in head and neck squamous cell carcinoma[J] Oncotarget,2015,6(1):101-115 doi:10. 18632/oncotarget .2684.
[18] Schmitt K,Molfenter B,Laureano NK,et al. Somatic mutations and promotor methylation of the ryanodine receptor 2 is a common event in thepathogenesis of head and neck cancer[J].Int J Cancer,2019,145(12):3299-3310 doi: 10.1002/jc 32481.
[19] Owusu OE ,Rusciano 1,Marvi MV,et al.Phosphoinositide-Dependent signaling in cancer:a focus on phospholipase C isozymes[J.Int J Mol Sci,2020. 21(7):2581 .doi:10 3390/ims21072581.